Nachdem ich in einem anderen Beitrag schon ein bisschen was darüber erzählt habe, wie man auf TikZ-Knoten in chemfig-Molekülen und -Schemata zugreifen kann, möchte ich jetzt auf einen anderen Aspekt eingehen.
chemfig stellt schon eine Reihe von Bindungstypen bereit, ein paar seltenere aber durchaus benötigte Varianten gibt es aber nicht. Zu nennen wären etwa delokalisierte Doppelbindungen oder »gewellte« Bindungen.
Natürlich sind gewellte Bindungen nicht wirklich gewellt. Durch die Wellenform wird je nach Gebrauch eben eine bestimmte Information vermittelt. Zum Beispiel sind sie mir in Kohlenhydraten untergekommen, wo man durch Wellenform der Bindung zum anomeren OH zu erkennen gibt, dass sowohl alpha- als auch beta-Anomer gemeint sind.
Nun stellt sich die Frage, wie man solche Bindungen mit chemfig darstellen kann.
Hier können wir die Tatsache ausnützen, dass es chemfig ermöglicht, TikZ-Optionen an Bindungen weiterzugeben. Eine Bindung hat fünf optionale Argumente, getrennt durch Kommas in eckigen Klammern:
Das bedeutet, wir können Bindungen im TikZ-Sinne »dekorieren«. Eine solche naheliegende Dekoration ist snake:
\usepackage{chemfig}
\usetikzlibrary{decorations.pathmorphing}
\begin{document}
\chemfig{C-[,,,,decorate,decoration=snake]C}
\end{document}
Das Ergebnis ist allerdings nicht wirklich optimal. Zum Glück für uns hat @Jake auf TeX.sx aber eine Dekoration namens complete sines definiert, bei der die Dekoration die komplette Länge des Pfades ausnützt ohne die komischen geraden Start- und Endstücke.
\usetikzlibrary{decorations,decorations.pathmorphing}
\pgfdeclaredecoration{complete sines}{initial}{
\state{initial}[
width=+0pt,
next state=sine,
persistent precomputation={
\pgfmathsetmacro\matchinglength{
\pgfdecoratedinputsegmentlength /
int(\pgfdecoratedinputsegmentlength/\pgfdecorationsegmentlength)
}
\setlength{\pgfdecorationsegmentlength}{\matchinglength pt}
}
]{}
\state{sine}[width=\pgfdecorationsegmentlength]{
\pgfpathsine{
\pgfpoint
{0.25\pgfdecorationsegmentlength}
{0.5\pgfdecorationsegmentamplitude}
}
\pgfpathcosine{
\pgfpoint
{0.25\pgfdecorationsegmentlength}
{-0.5\pgfdecorationsegmentamplitude}
}
\pgfpathsine{
\pgfpoint
{0.25\pgfdecorationsegmentlength}
{-0.5\pgfdecorationsegmentamplitude}
}
\pgfpathcosine{
\pgfpoint
{0.25\pgfdecorationsegmentlength}
{0.5\pgfdecorationsegmentamplitude}
}
}
\state{final}{}
}
Damit sieht das schon viel besser aus. \chemfig{C-[,,,,decorate,decoration=complete sines]C}:
Nun kann man sich damit zum Beispiel einen TikZ-Stil definieren und etwa noch Amplitude und Anzahl der Perioden pro Bindung einstellen:
\tikzset{
wavy bond/.style =
{
decorate,
decoration =
{
complete sines,
amplitude = 0.3em,
% chemfigs Bindungslänge:
segment length =
\the\dimexpr\csname CF@atom@sep\endcsname/5\relax
}
}
}
Joseph Wright hat diese Idee in seinem Blog übrigens auch schon aufgegriffen. Damit wird dann \chemfig{-[,,,,wavy bond]}
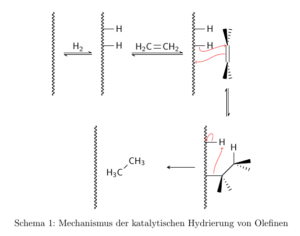
Um zum Schluss noch ein nettes Bild zu bekommen, verwenden wir jetzt die neue Dekoration und die Technik aus dem schon erwähnten letzten Beitrag, um die oberflächenkatalysierte Hydrierung von Olefinen zu demonstrieren:
\documentclass{scrartcl}
\usepackage[ngerman]{babel}
\usepackage[ghsystem=false]{chemmacros}
\usepackage{chemfig}
% bei Joseph Wright geklaut: http://www.texdev.net/tag/chemfig/
\setdoublesep{0.35700 em} % 'Bond Spacing'
\setatomsep{1.78500 em} % 'Fixed Length'
\setbondoffset{0.18265 em} % 'Margin Width'
\newcommand{\bondwidth}{0.06642 em} % 'Line Width'
\setbondstyle{line width = \bondwidth}
\newcommand*{\bondboldwidth}{0.22832 em} %'Bold Width'
\newcommand*{\bondhashlength}{0.25737 em} % 'Hash Spacing'
\setcrambond
{\the\dimexpr \bondwidth * 2 + \bondboldwidth \relax}
{\bondwidth}{\bondhashlength}
\setstacksep{.7ex}
\usetikzlibrary{decorations,decorations.pathmorphing}
\pgfdeclaredecoration{complete sines}{initial}{
\state{initial}[
width=+0pt,
next state=sine,
persistent precomputation={
\pgfmathsetmacro\matchinglength{
\pgfdecoratedinputsegmentlength /
int(\pgfdecoratedinputsegmentlength/\pgfdecorationsegmentlength)
}
\setlength{\pgfdecorationsegmentlength}{\matchinglength pt}
}
]{}
\state{sine}[width=\pgfdecorationsegmentlength]{
\pgfpathsine{
\pgfpoint
{0.25\pgfdecorationsegmentlength}
{0.5\pgfdecorationsegmentamplitude}
}
\pgfpathcosine{
\pgfpoint
{0.25\pgfdecorationsegmentlength}
{-0.5\pgfdecorationsegmentamplitude}
}
\pgfpathsine{
\pgfpoint
{0.25\pgfdecorationsegmentlength}
{-0.5\pgfdecorationsegmentamplitude}
}
\pgfpathcosine{
\pgfpoint
{0.25\pgfdecorationsegmentlength}
{0.5\pgfdecorationsegmentamplitude}
}
}
\state{final}{}
}
\tikzset{
bold bond/.style = {line width = \bondboldwidth},
dash bond/.style =
{dash pattern = on \bondhashlength off \bondhashlength},
hash bond/.style =
{
dash pattern = on \bondwidth off \bondhashlength,
line width = \bondboldwidth
},
wavy bond/.style =
{
decorate,
decoration =
{
complete sines,
amplitude = \bondboldwidth,
% chemfigs Bindungslänge:
segment length =
\the\dimexpr\csname CF@atom@sep\endcsname/5\relax
}
}
}
\renewcommand*\printatom[1]{\ensuremath{\mathsf{#1}}}
\setchemformula{format=\sffamily}
\DeclareNewTOC[
type=scheme,
types=schemes,% used in the \listof.. command
float,
name=Schema,
listname={Verzeichnis der Schemata}
]{los}
\newcommand*\elmove[4]{%
\chemmove{
\draw[red,->,shorten >=2pt,shorten <=2pt]
(#1) .. controls +(#2) and +(#4) .. (#3) ;
}%
}
\definesubmol\wave{-[,,,,wavy bond]}
\begin{document}
\begin{scheme}
\centering
\schemestart
\chemfig{[6]!\wave!\wave!\wave!\wave!\wave}
\arrow{<=>[\ch{H2}]}
\chemfig{[6]!\wave(-[0]H)!\wave(-[0]H)!\wave!\wave!\wave}
\arrow{<=>[\chemfig{H_2C=CH_2}]}[,1.5]
\chemfig{[6]!\wave(-[0]H)!\wave(-[@{b1}0]H)!\wave @{kat1}!\wave!\wave}
\arrow{0}[,.2]
\chemfig{>[:-80]@{C}(<:[:80])=[@{db}6](<:[:-80])<[:-100]}
\arrow{<=>}[-90]
\chemfig{
[6]!\wave @{kat2}%
(-[@{b2}0]@{H}H)
!\wave!\wave
(-[@{b3}0](<[:-100])(<:[:-60])-[1](<[:10])(<:[:-20])-[2]H)
!\wave!\wave
}
\arrow[180]
\subscheme{
\chemfig{[6]!\wave!\wave!\wave!\wave!\wave}
\arrow{0}[,.2]
\chemfig{H_3C-[1]CH_3}
}
\elmove{b1}{-90:5mm}{C}{180:5mm}
\elmove{db}{180:7mm}{kat1}{0:7mm}
\elmove{b2}{70:5mm}{kat2}{70:5mm}
\elmove{b3}{90:5mm}{H}{-90:5mm}
\schemestop
\caption{Mechanismus der katalytischen Hydrierung von Olefinen}
\end{scheme}
\end{document}

Comments
2 responses to “Gewellte Bindungen – chemfig”
Den Stil für die komplette decoration finde ich gerade besonders nützlich! Ich habe schon mehrfach wegen der Randstücke bei Wellenlinien an Parametern gedreht oder workarounds gemacht. So wie in Deinem Artikel geschrieben und von Jake vorgeschlagen ist es richtig gut.
Stimmt, die
complete sinesdecoration lässt sich natürlich ohne weiteres auch für andere Zwecke einsetzen.